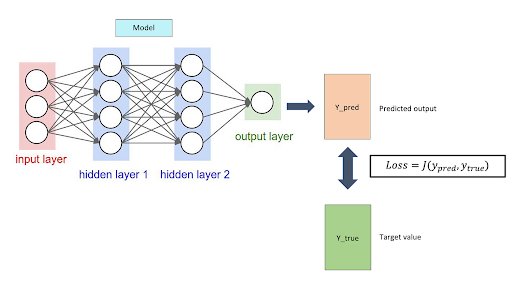
Réseaux de neurones
Perceptron, Fast forward
- perceptron
- multi layer perceptron
- backpropagation
- fonctions d’activation
- keras, tensorflow, pytorch, jax
- régularisation
- dropout
- batch normalisation
Fonction de cout
Quelques dates
- 1943 : neurone artificiel (McCulloch & Pitts)
- 1957 : perceptron de Rosenblatt + apprentissage
- 1969 : limites du perceptron (Minsky & Papert)
- Années 1980 : backpropagation
Article : Réseaux de neurones sur Wikipedia
A la base, le neurone
- regression lineaire ou les coefficients sont les poids + un biais
- et une fonction d’activation
Équivalent à une régression linéaire.
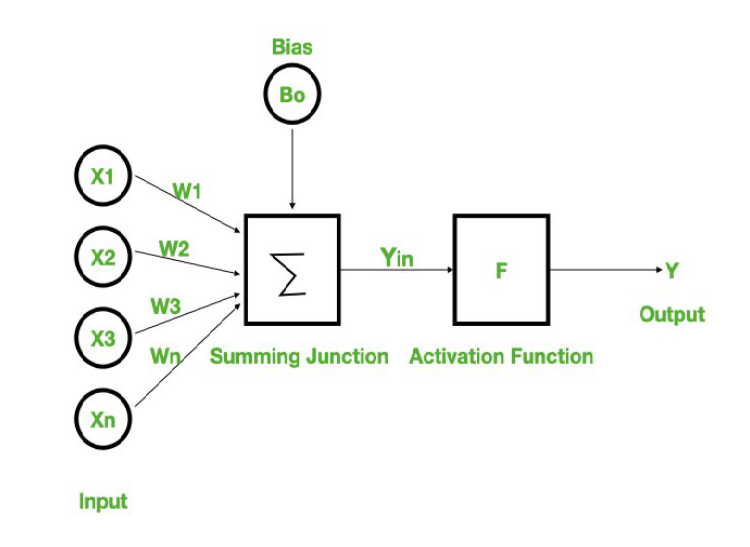
y = F(x1 .w1 + x2.w2 + ... + xN.wN + b)
fonction d’activation : analogie avec les neurones du cerveau; capacité à apprendre des relations non linéaires a partir d’une relation lineaire.
Le choix de la fonction d’activation va influencer le comportement du reseaux et notamment contret sa tendance à exploser (exploding gradient) ou a disparaitre (vanishing gradient). Et surtout sa rapidité de calcul.
Contexte d’une classification binaire linéairement séparable
- des échantillons de données de dimension N
- coefficients à apprendre (N), taux_apprentissage (learning rate), seuil de classification
en python
import numpy as np
# Données
X = np.array([[0, 0], [0, 1], [1, 0], [1, 1]])
# Example linearement separable
y = np.array([1, 0, 1, 0])
# XOR : non linearement separable
# y = np.array([0, 1, 1, 0])
# Initialisation
coefs = np.zeros(2)
bias = 0
taux_apprentissage = 0.2
# Entraînement
for epoch in range(20):
# pour chaque échantillon (choisi aléatoirement)
for idx in np.random.choice(len(X), len(X)):
x = X[idx]
# produit scalaire + biais => output de la regression lineaire
valeur = np.dot(x, coefs) + bias
# fonction activation (heavyside)
prediction = 1 if valeur > 0.5 else 0
# le perceptron n'apprend que de ses erreurs
# mettre a jour coefs / biais si prediction != vraie valeur
if prediction != y[idx]:
erreur = y[idx] - prediction
coefs += taux_apprentissage * erreur * x
bias += taux_apprentissage * erreur
print("epoch", epoch, prediction == y[idx], "coefs", coefs, "bias", bias)
limites du percetron une couche
- Un perceptron à une seule couche ne peut pas apprendre des fonctions non linéaires, malgré le fait que la fonction d’activation (fonction de Heaviside) soit non linéaire.
- Architecture trop simple: pas de couches cachées qui sont nécessaires pour capturer des relations non linéaires entre les entrées et la sortie.
- Pour apprendre des fonctions non linéaires, des architectures de réseaux de neurones plus avancées sont utilisées. Par exemple les perceptrons multicouches avec des couches cachées associées à des fonctions d’activation non linéaires,.
Different shades of Le percetron
Single cell (1 neurone):
- Classification binaire (0 ou 1)
- Sépare 2 classes seulement
Single layer avec plusieurs cellules:
- Classification multi-classe (3, 4, 5… classes)
- Chaque neurone = une classe
Exemple : 3 neurones pour reconnaître A, B, C
- Mais chaque neurone voit les données brutes directement
- Toujours linéaire séparable
Multi-layer:
- Couches cachées intermédiaires
- Les couches cachées créent des features complexes
- Peut résoudre le XOR et problèmes non-linéaires
XOR vs AND
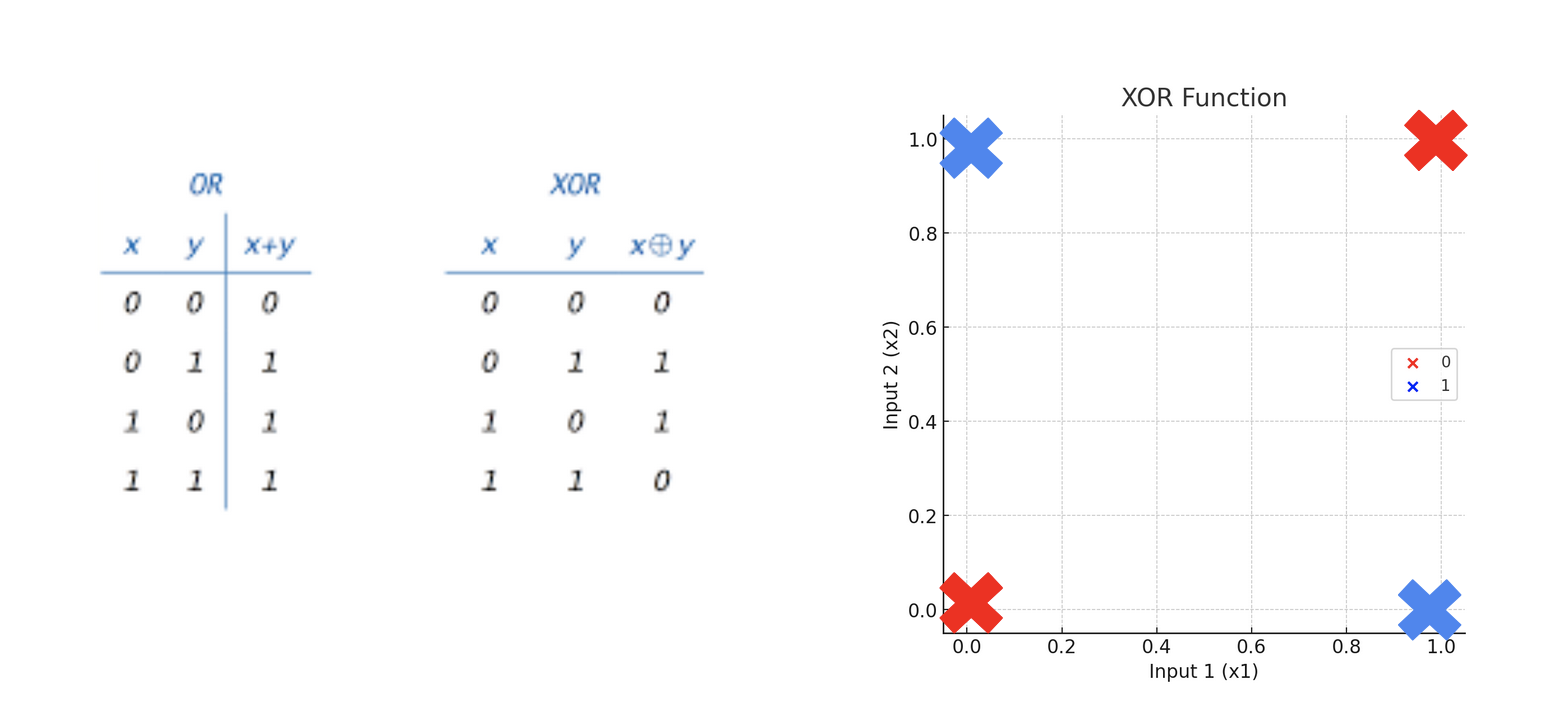
XOR / AND - Notebook
Notebook: XOR vs AND
- code du perceptron
- version basique
- puis ajouter le learning rate
- Demo OR, XOR
- influence de l’initialisation des poids et du biais
- generer des blobs
- séparable et non-séparable
- experimenter avec
- learning rate
- epochs
Multi layer perceptron
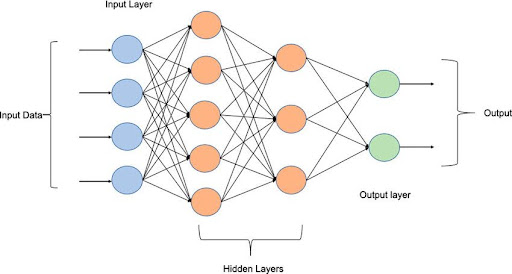
MLP — Propriétés
- apprend des fonctions non-linéaires
- plusieurs minimums locaux → sensible à l’initialisation
- tuning hyperparamètres:
- couches, cellules / noeuds, learning rate, epochs
- nécessite normalisation
MLP avec scikit-learn

hidden_layer_sizes=(100,): The size of the hidden layersactivation='relu': The activation function for the hidden layerssolver='adam': The solver for weight optimizationalpha=0.0001: Strength of the L2 regularization termbatch_size='auto': Size of minibatches for stochastic optimizers
learning rate :
learning_rate='constant': Constant learning ratelearning_rate_init=0.001: The initial learning rate usedpower_t=0.5: The exponent for inverse scaling learning rate
optimisation :
max_iter=200: Maximum number of iterationsshuffle=True: Whether to shuffle training data before training
et
random_state=None: The seed of the pseudo random number generatortol=0.0001: Tolerance for the optimizationverbose=False: Whether to print progress messages to stdoutwarm_start=False: Whether to reuse the solution of the previous call to fit as initializationvalidation_fraction=0.1: The fraction of training data to set aside as validation set for early stoppingearly_stopping=False: Whether to use early stopping to terminate training when validation score is not improvingn_iter_no_change=10: Maximum number of epochs to not meet tol improvement
solvers:
momentum=0.9: Momentum for gradient descent, Only used when solver=’sgd’.nesterovs_momentum=True: Nesterov’s momentum, Only used when solver=’sgd’.beta_1=0.9: Coefficient used for computing running averages of gradient, Only used when solver=’adam’.-
beta_2=0.999: Coefficient used for computing running averages of square of gradient, Only used when solver=’adam’. epsilon=1e-08: Value for numerical stability. Only used when solver=’adam’.max_fun=15000: Maximum number of loss function calls. Only used when solver=’lbfgs’.
Backpropagation
nécessaire pour adapter les coefficients des noeuds en fonction de l’erreur et du résultat de la fonction de coût
pass forward
- input data est passée de couche en couche de gauche à droite,
- chaque couche calcule une prédiction, une valeur à travers la fonction d’activation
- en sortie du réseau, calcul de l’écart entre prédiction et la valeur réelle avec la fonction de coût
backward
- l’erreur est propagée dans chaque noeud de droite à gauche et le gradient de la fonction de coût est calculé pour chaque noeud
- les coeffs sont maj en utilisant le gradient
C’est comme pour le gradient descent, mais en plusieurs couches.
Impact de la derivee de la fonction d’activation sur la vitesse d’apprentissage
gradient = dérivée
Pour un neurone unique
Considérons un neurone simple avec :
- Entrée : x
- Poids : w
- Biais : b
- Somme pondérée : z = wx + b
- Fonction d’activation : f
- Sortie : y = f(z)
Lors de la backpropagation, on veut calculer comment ajuster le poids w pour minimiser l’erreur. Pour cela, on a besoin de ∂L/∂w le gradient (aka la dérivée) de la loss function L par rapport à w.
w_{t+1} = w_{t} - learning_rate * ∂L/∂w
En utilisant la règle de dérivation en chaîne :
∂L/∂w = ∂L/∂y × ∂y/∂z × ∂z/∂w
Or :
- ∂L/∂y est le gradient qui arrive depuis la couche suivante (voir plus loin)
- ∂y/∂z = f’(z) est la dérivée de la fonction d’activation
- ∂z/∂w = x
Donc : ∂L/∂w = ∂L/∂y × f’(z) × x
Sans f’(z), on ne peut pas calculer le gradient ! La dérivée agit comme un “multiplicateur” qui module l’amplitude du gradient qui se propage.
Pour une couche complète
Pour une couche avec plusieurs neurones, chaque neurone i a sa propre sortie y_i = f(z_i).
Le gradient qui arrive de la couche suivante (∂L/∂y) doit être propagé vers la couche précédente. Pour chaque neurone i de la couche :
∂L/∂z_i = ∂L/∂y_i × f'(z_i)
La dérivée f’(z_i) détermine donc :
-
L’amplitude de propagation : Si f’(z_i) ≈ 0 (zones saturées de sigmoid/tanh), le gradient devient presque nul → problème de “vanishing gradient”
-
La direction d’ajustement : Le signe de f’(z_i) influence si on augmente ou diminue les poids
-
La vitesse d’apprentissage locale : Une grande valeur de f’(z_i) amplifie le gradient, permettant des ajustements plus importants
gradient qui arrive depuis la couche suivante
exemple sur architecture simple à 3 couches
Imaginons un réseau très simple :
Entrée (x) → Couche 1 → Couche 2 → Couche 3 (sortie) → Perte (L)
Avec les notations :
- Couche 1 : sortie = y₁
- Couche 2 : sortie = y₂
- Couche 3 : sortie = y₃
- Perte : L = fonction_perte(y₃, cible)
Le flux de la backpropagation: La backpropagation calcule les gradients en partant de la fin (de la perte) et en remontant vers le début :
L ← y₃ ← y₂ ← y₁ ← x
Étape 1 : On part de la perte
D’abord, on calcule ∂L/∂y₃ (comment la perte change quand la sortie finale change)
= dérivée de la fonction de cout / y₃
Étape 2 : On remonte à la couche 2
Pour mettre à jour les poids de la couche 2, on a besoin de ∂L/∂y₂
Mais la perte L ne dépend pas directement de y₂. Elle dépend de y₂ à travers y₃ :
L dépend de y₃
y₃ dépend de y₂
Donc L dépend de y₂
Par la règle de la chaîne :
∂L/∂y₂ = ∂L/∂y₃ × ∂y₃/∂y₂
∂L/∂y₃ est le “gradient qui arrive depuis la couche suivante” (couche 3)
Pourquoi “arrive depuis la couche suivante” ?
Quand on est à la couche 2 et qu’on veut calculer nos gradients locaux :
- On ne recalcule pas tout depuis la perte L
- On reçoit ∂L/∂y₂ qui a déjà été calculé par la couche 3
- Ce gradient nous dit : “Si tu changes ta sortie y₂ d’une petite quantité, voici comment ça affectera la perte finale L”
Code conceptuel
# Forward pass
y1 = couche1(x)
y2 = couche2(y1)
y3 = couche3(y2)
L = perte(y3, cible)
# Backward pass
grad_y3 = ∂L/∂y3 # Calculé directement depuis la perte = dérivée de la fonction de Loss
# Pour la couche 2
grad_y2 = grad_y3 × ∂y3/∂y2 # Le gradient "arrive" de la couche 3
# Maintenant on peut utiliser grad_y2 pour mettre à jour les poids de couche2
# Pour la couche 1
grad_y1 = grad_y2 × ∂y2/∂y1 # Le gradient "arrive" de la couche 2
C’est cette propagation en cascade des gradients qui donne son nom à la “back-propagation” : les gradients se propagent de l’arrière vers l’avant du réseau.
Exemple avec des chiffres : a-step-by-step-backpropagation-example
Fonctions d’activation
- sigmoid [0,1] → problème gradient vanishing, pas centrée
- tanh [-1,1] → calcul coûteux
- ReLU [0,∞] → simple, rapide, favorise convergence (souvent le choix)
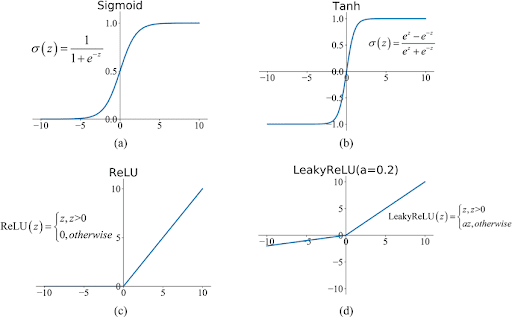
Chaque fonction introduit une non-linéarité différente qui impact la vitesse d’apprentissage et la convergence.
Certaines fonctions ont des zones “mortes” (dead zone) où rien n’est plus appris (ReLU : zone négative)
Sigmoid
- Lisse et différentiable partout
- Problème : gradient très petit aux extrémités → apprentissage lent (vanishing gradient)
- Bon pour la couche de sortie en classification binaire
ReLU (Rectified Linear Unit)
- Sortie : max(0, x)
- Rapide à calculer
- Apprend plus vite que Sigmoid
- Problème : zone négative “morte” (gradient = 0 si x < 0)
- Standard pour les couches cachées
Tanh
- Sortie entre -1 et 1
- Meilleur que Sigmoid, updates plus rapides. Le gradient est plus fort pour tanh que pour sigmoid (derivée en 0 = 1, derivée en 0 pour sigmoid = 0.25))
- Mais aussi plus lent à calculer, plus instable
Softmax
- Convertit les valeurs en probabilités (somme = 1)
- Utilisé pour multi-classe en derniere couche de sortie
Leaky ReLU
- Comme ReLU mais avec une petite pente négative
- Évite le problème des zones mortes
Zones mortes
Les zones mortes en ReLU = les neurones ne s’entraînent plus
def ReLU(x):
return max(0, x)
- Si x > 0 : la fonction retourne x, le gradient = 1 ✅ (apprend)
- Si x < 0 : la fonction retourne 0, le gradient = 0 ❌ (n’apprend plus)
Donc si un neurone reçoit toujours des valeurs négatives :
- Il sort toujours 0
- Le gradient est 0
- Les poids ne changent jamais
- Le neurone est “mort”, il n’apporte plus d’info au réseau
La solution :
- Leaky ReLU : au lieu de 0, retourner 0.01 * x si x < 0
- Ainsi le gradient n’est jamais vraiment 0, les neurones peuvent continuer à apprendre
vanishing gradient
le probleme avec la sigmoide et la tanh est que le gradient est très petit aux extrémités.
Comme on multiple par la derivée en x de couche en couche il y a propagation de la multiplication par une valeur petite => la maj devient super petite et le gradient disparaît
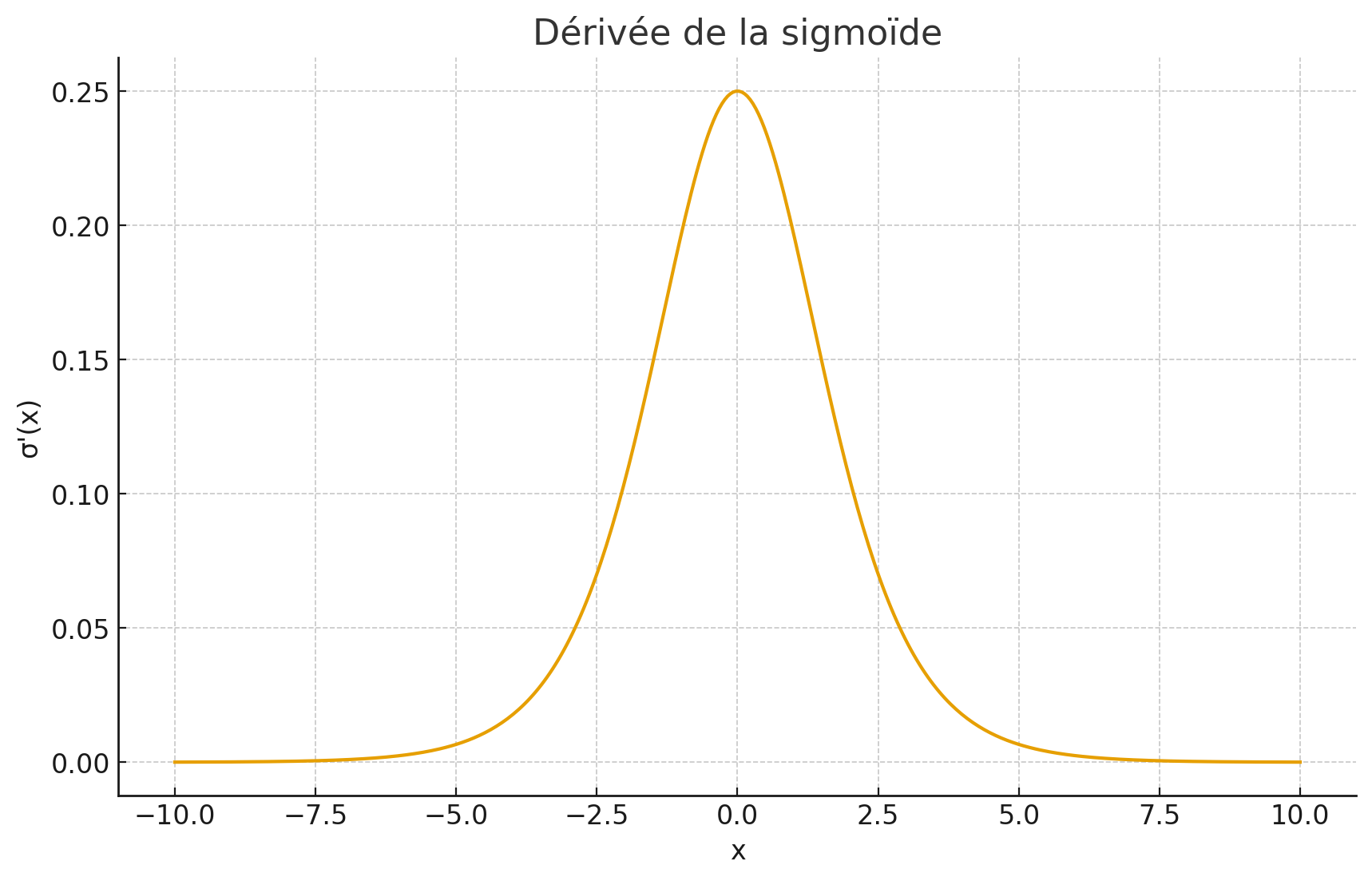
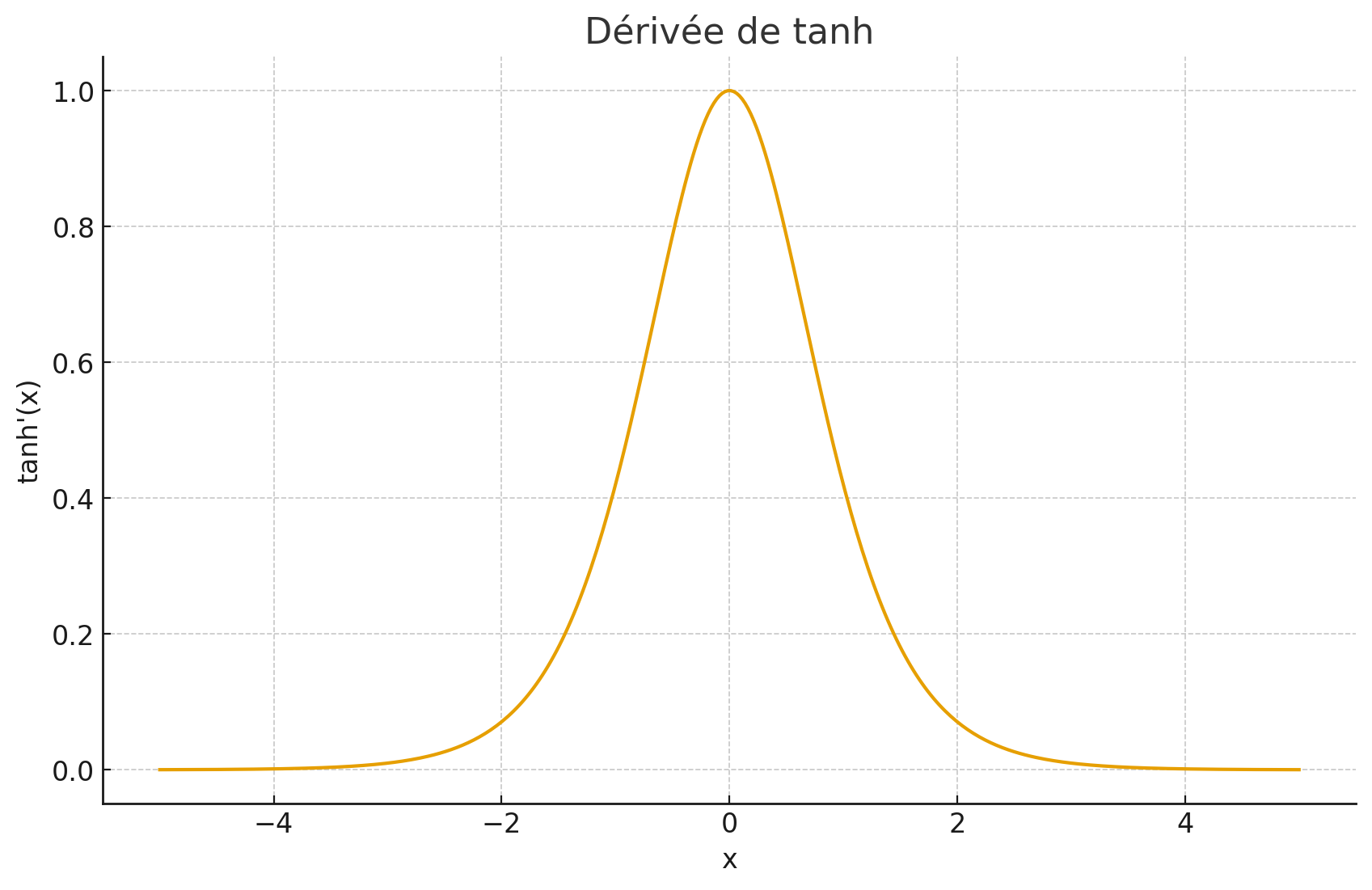
Vanishing Gradient = les gradients deviennent de plus en plus petits en remontant dans le réseau.
Pourquoi avec Sigmoid ?
Sigmoid a une dérivée max de 0.25. Quand on a plusieurs couches :
Couche 1: gradient = 0.25
Couche 2: gradient = 0.25 * 0.25 = 0.0625
Couche 3: gradient = 0.0625 * 0.25 = 0.015625
Couche 4: gradient = 0.015625 * 0.25 = 0.004
Chaque couche multiplie par 0.25, ça devient exponentiellement petit.
Problème:
- Les couches au début du réseau (a gauche) reçoivent des gradients quasi nuls
- Elles n’apprennent presque rien
- L’entraînement devient très lent ou s’arrête
Exemple visuel:
Sortie: gradient = 0.5
Couche cachée 5: 0.5 * 0.25 = 0.125
Couche cachée 4: 0.125 * 0.25 = 0.03
Couche cachée 3: 0.03 * 0.25 = 0.007
Couche cachée 1: ~ 0.000001 ← QUASI MORT
Solutions:
- Utiliser ReLU (dérivée = 1, pas de multiplication)
- Batch normalization
- Skip connections (ResNets)
C’est pourquoi ReLU domine aujourd’hui ! 🎯
Exploding gradient
Inversement on a le probleme des gradients qui explosent
Le problème de l’exploding gradient
Gradient final = gradient_initial × f'(z₁) × W₁ × f'(z₂) × W₂ × ... × f'(zₙ) × Wₙ
Si ces valeurs sont grandes (ex: |W| > 1 et f’(z) ≈ 1), après 10 couches :
- 1.5¹⁰ ≈ 58
- 2¹⁰ = 1024
- 3¹⁰ ≈ 59,000 !
Conséquences concrètes :
- Les poids font des “bonds gigantesques” au lieu de petits ajustements
- Le modèle oscille violemment sans converger
- L’entraînement devient instable ou échoue complètement
Techniques de remédiation du exploding gradient
- 1) Gradient Clipping (le plus simple)
On “coupe” les gradients trop grands quand ils depassent un certains seuil. Simple et efficace
if |gradient| > seuil:
gradient = +/- seuil
- 2) Initialisation appropriée des poids
- Initialiser les poids avec des valeurs plus petites pour que les multiplications n’explosent pas.
- 3) choisir une fonction d’activation qui a une dérivée qui est constante
- Éviter sigmoid/tanh dans les réseaux très profonds (dérivées peuvent amplifier)
- Préférer ReLU et variantes : dérivée = 0 ou 1, pas d’amplification
- ELU, SELU : auto-normalisantes
- 4) Réduction du learning rate
Voir aussi
- batch normalization: on normalize la sortie des fonctions d’activation
- ResNet : on saute certaines couches
Optimizers (solvers)
- Backpropagation met à jour les poids selon le gradient de la loss.
- SGD vs Adam vs RMSprop
Les solvers = optimiseurs qui ajustent les poids pendant l’entraînement.
SGD (Stochastic Gradient Descent)
- Le plus simple :
W = W - learning_rate * gradient - Lent mais stable
- Usage : modèles simples, quand tu as du temps
Momentum
- Ajoute de l’inertie (comme une luge qui descend la pente et qui traine un poids)
W = W - learning_rate * gradient + momentum * previous_step- Plus rapide que SGD
- Usage : accélère la convergence
RMSprop
- Adapte le learning rate par paramètre
- Divise le gradient par sa moyenne mobile
- Bon pour les gradients instables
- Usage : réseaux de neurones moyens
Adam ⭐ (le plus frequent)
- Combine Momentum + RMSprop
- Adaptatif et rapide
W = W - learning_rate * (momentum * gradient) / (sqrt(variance) + eps)- Usage : presque tout! C’est souvent le choix par défaut
Différences principales:
| Solver | Vitesse | Stabilité | Mémoire |
|---|---|---|---|
| SGD | Lent | Très stable | Faible |
| Momentum | Rapide | Stable | Faible |
| RMSprop | Rapide | Bon | Faible |
| Adam | Très rapide | Très bon | Moyen |
Comment choisir ?
- Commencer avec Adam → marche bien partout
- SGD → si tu veux la meilleure généralisation (overfit moins)
- RMSprop → si Adam cause des problèmes
- Momentum → rarement, Adam est mieux
Règle simple: Adam d’abord, puis ajuste si besoin !
Atelier MLP — MNIST
- 28x28 images de chiffres (0–9)
- 60k train / 10k test
- tuning hyperparamètres
- classification binaire ou multiclasse
Refs:
Régularisation : limiter l’overfit sans ajouter de biais
Ajouter une contrainte pour empêcher le modèle de coller trop aux données d’entraînement.
Contraintes possibles :
- L1, L2 sur la fonction de coût
- réduire complexité (# couches / # neurones)
Arbres de décision :
- limiter profondeur
- min samples / leaf
- bagging (forêts aléatoires)
Régularisation des réseaux de neurones
- L1/L2 Regularization
- réduire complexité du reseau : moins de couches, moins de neurones
- ajouter du bruit aux données
Dropout
- annule aléatoirement des neurones (ex: p=0.2)
- simple & efficace, force le réseau à être redondant
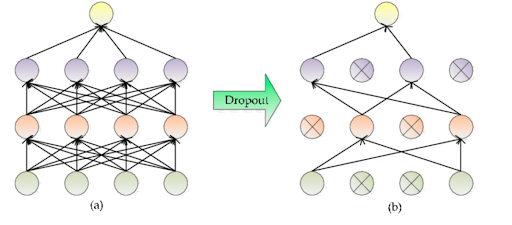
Batch normalization
- Normalise les activations entre couches
- Stabilise l’entraînement et réduit l’overfitting
-
Usage : standard dans les réseaux modernes
- Dropout = le plus simple et efficace
- L2 = léger, facile à implémenter
- Early Stopping = indispensable
- Batch Norm = bonus, améliore tout
En pratique: Dropout + Early Stopping = 90% du travail !
Resume
- une cellule = regression lineaire + fonction activation
- MLP = plusieurs couches de neurones, capable de représenter des relations non linéaires
- fonction d’activation : ReLU (importance de la derivée)
- solver : Adam
- regularisation : L2 / Dropout / BatchNorm / Early Stopping